Fw: [求救] 使用Galaxy無法input reference genome
看板BioMedInfo (生醫資訊)作者keroromoa (夏天一到就凸顯自己很魯)時間8年前 (2017/08/22 13:25)推噓0(0推 0噓 1→)留言1則, 1人參與討論串1/2 (看更多)
※ [本文轉錄自 Biotech 看板 #1Pcy0DZb ]
作者: keroromoa (夏天一到就凸顯自己很魯) 看板: Biotech
標題: [求救] 使用Galaxy無法input reference genome
時間: Tue Aug 22 13:24:24 2017
小弟目前正在使用Galaxy proteogenomics進行分析測試,
由於不確定Galaxy能夠input多大的data,
所以目標是在實驗室架設主機,將Galaxy tutorial的workflow複製到實驗室的server
http://js-157-167.jetstream-cloud.org/ (需要註冊才能查看tutorial workflow)
將workflow所需要的tool全部都安裝完後,
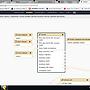
發現大多數的tool在select a reference genome(annotation)全都顯示空白,如附圖所示
紅框中,Galaxy主機在各個tool的reference genome(annotation)中都有出現相關的索引
http://imgur.com/NoNNgFe



綠框中,實驗室的主機在各個tool的reference genome(annotation)中都呈現空白
http://imgur.com/gahVZAX



接著參考這個教學,https://goo.gl/QCxU9e,用data manager來建立built-in genomes
可是按照教學先從UCSC/NCBI存在的資料庫import進去,
不管怎麼修改DBKey或accession number都是無限error
http://imgur.com/jCHi0Xt

於是自己從UCSC下載hg38 fasta file,用new的方式import
http://imgur.com/Tm54uhG

雖然成功了,但是workflow相關的tool(Bowtie2、BWA)卻依舊No options available
http://imgur.com/YipCPjW


在只能用遠端連線而無法動主機在linux上輸入指令的情況下,
請問各位前輩還有什麼方法能解決嗎?
雖然裡面有說請直接聯繫Galaxy,
可是那個意思看起來比較像如果題目較冷門找不到reference genome可去聯繫,
不過我想要的reference genome只是很基本的human GRCh38(hg38)啊...謝謝大家~
--
※ 發信站: 批踢踢實業坊(ptt.cc), 來自: 163.25.93.81
※ 文章網址: https://www.ptt.cc/bbs/Biotech/M.1503379469.A.8E5.html
※ 發信站: 批踢踢實業坊(ptt.cc)
※ 轉錄者: keroromoa (163.25.93.81), 08/22/2017 13:25:18
→
08/22 22:11, , 1F
08/22 22:11, 1F
討論串 (同標題文章)
BioMedInfo 近期熱門文章
PTT職涯區 即時熱門文章